Riferimenti >> Spettrometria di Massa
Spettrometria di Massa
la spettrometria di Massa è una potente tecnica analitica utilizzata per quantificare noto materiali, per identificare composti sconosciuti all’interno di un campione, e a chiarire la struttura e proprietà chimiche delle molecole., Il processo completo comporta la conversione del campione in ioni gassosi, con o senza frammentazione, che sono poi caratterizzati dal loro rapporto massa/carica (m / z) e abbondanze relative.
Questa tecnica studia fondamentalmente l’effetto dell’energia ionizzante sulle molecole. Dipende dalle reazioni chimiche nella fase gassosa in cui le molecole del campione sono consumate durante la formazione di specie ioniche e neutre.,
Principio di base
Uno spettrometro di massa genera più ioni dal campione in esame, quindi li separa in base al loro specifico rapporto massa-carica (m / z) e quindi registra l’abbondanza relativa di ciascun tipo di ioni.
Il primo passo nell’analisi spettrometrica di massa dei composti è la produzione di ioni in fase gassosa del composto, fondamentalmente per ionizzazione elettronica. Questo ion molecolare subisce la frammentazione. Ogni ion prodotto primario derivato dallo ion molecolare, a sua volta, subisce la frammentazione, e così via., Gli ioni sono separati nello spettrometro di massa in base al loro rapporto massa-carica e vengono rilevati in proporzione alla loro abbondanza. Viene quindi prodotto uno spettro di massa della molecola. Visualizza il risultato sotto forma di un diagramma di abbondanza di ioni rispetto al rapporto massa-carica. Gli ioni forniscono informazioni riguardanti la natura e la struttura della loro molecola precursore. Nello spettro di un composto puro, lo ion molecolare, se presente, appare al valore più alto di m/z (seguito da ioni contenenti isotopi più pesanti) e dà la massa molecolare del composto.,
Componenti
Lo strumento è costituito da tre componenti principali:
-
Sorgente ionica: Per la produzione di ioni gassosi dalla sostanza studiata.
-
Analizzatore: Per risolvere gli ioni nelle loro caratteristiche componenti di massa in base al loro rapporto massa-carica.
-
Sistema rivelatore: Per rilevare gli ioni e registrare l’abbondanza relativa di ciascuna delle specie ioniche risolte.,
Inoltre, è necessario un sistema di introduzione del campione per ammettere i campioni da studiare alla sorgente ionica mantenendo i requisiti di alto vuoto (~10-6 a 10-8 mm di mercurio) della tecnica; ed è necessario un computer per controllare lo strumento, acquisire e manipolare i dati e confrontare gli spettri con le librerie di riferimento.,
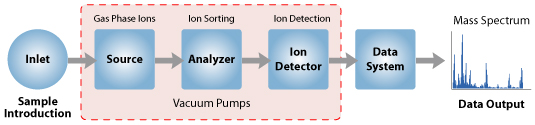
Con tutti i componenti di cui sopra, uno spettrometro di massa deve sempre eseguire i seguenti processi:
-
Produrre ioni dal campione nella sorgente di ionizzazione.
-
Separare questi ioni in base al loro rapporto massa-carica nell’analizzatore di massa.
-
Infine, frammentare gli ioni selezionati e analizzare i frammenti in un secondo analizzatore.,
-
Rileva gli ioni che emergono dall’ultimo analizzatore e misura la loro abbondanza con il rilevatore che converte gli ioni in segnali elettrici.
-
Elaborare i segnali dal rivelatore che vengono trasmessi al computer e controllare lo strumento utilizzando feedback.
L’analisi delle biomolecole mediante spettrometria di massa
La spettrometria di massa sta rapidamente diventando un campo indispensabile per l’analisi delle biomolecole., Fino al 1970, le uniche tecniche analitiche che fornivano informazioni simili erano i metodi elettroforetici, cromatografici o ultracentrifugazione. I risultati non sono stati assoluti in quanto basati su caratteristiche diverse dal peso molecolare. Così l’unica possibilità di conoscere il peso molecolare esatto di una macromolecola è rimasto il suo calcolo basato sulla sua struttura chimica.,
Lo sviluppo di metodi di ionizzazione di desorbimento basati sull’emissione di ioni preesistenti come il desorbimento del plasma (PD), il bombardamento atomico veloce (FAB) o il desorbimento laser (LD), ha permesso l’applicazione della spettrometria di massa per l’analisi di biomolecole complesse.
Analisi dei glicani
Gli oligosaccaridi sono molecole formate dall’associazione di diversi monosaccaridi
legati attraverso legami glicosidici. La determinazione della struttura completa degli oligosaccaridi è più complessa di quella delle proteine o degli oligonucleotidi., Comporta la determinazione di componenti aggiuntivi come conseguenza della natura isomerica dei monosaccaridi e della loro capacità di formare oligosaccaridi lineari o ramificati. Conoscere la struttura di un oligosaccaride richiede non solo la determinazione della sua sequenza monosaccaridica e il suo modello di ramificazione, ma anche la posizione dell’isomero e la configurazione anomerica di ciascuno dei suoi legami glicosidici.
I progressi nella glicobiologia comportano uno studio completo della struttura, della bio-sintesi e della biologia di zuccheri e saccaridi., La spettrometria di massa (MS) sta emergendo come una tecnologia abilitante nel campo della glicomica e della glicobiologia.
Analisi dei lipidi
I lipidi sono costituiti da molte classi di molecole diverse che sono solubili in solventi organici. La lipidomica, una parte importante della metabolomica, costituisce l’analisi dettagliata e la caratterizzazione globale, sia spaziale che temporale, della struttura e della funzione dei lipidi (il lipidoma) all’interno di un sistema vivente.
Sono state sviluppate molte nuove strategie per analisi basate sulla spettrometria di massa dei lipidi., Le metodologie di lipidomica più diffuse comprendono sorgenti di ionizzazione elettrospray (ESI) e analizzatori a triplo quadrupolo. Utilizzando la spettrometria di massa, è possibile determinare il peso molecolare, la composizione elementare, la posizione di ramificazione e la natura dei sostituenti nella struttura lipidica.
Analisi di Proteine e peptidi
Proteine e peptidi sono polimeri lineari costituiti da combinazioni dei 20 amminoacidi legati da legami peptidici. Le proteine subiscono diverse modifiche post-traslazionali, estendendo la gamma della loro funzione tramite tali modifiche.,
Il termine Proteomica si riferisce all’analisi del contenuto proteico completo in un sistema vivente, incluse proteine co – e post-traduzionalmente modificate e varianti alternativamente impiombate. La spettrometria di massa è diventata una tecnica cruciale per quasi tutti gli esperimenti di proteomica. Permette la determinazione precisa della massa molecolare dei peptidi come pure delle loro sequenze. Queste informazioni possono essere utilizzate molto bene per l’identificazione delle proteine, il sequenziamento de novo e l’identificazione delle modifiche post-traduzionali.,
Analisi di oligonucleotidi
Oligonucleotidi (DNA o RNA), sono polimeri lineari di nucleotidi. Questi sono composti da una base azotata, uno zucchero ribosio e un gruppo fosfato. Gli oligonucleotidi possono subire diverse modificazioni covalenti naturali che sono comunemente presenti in tRNA e rRNA, o innaturali derivanti da reazioni con composti esogeni. La spettrometria di massa svolge un ruolo importante nell’identificare queste modifiche e determinare la loro struttura e la loro posizione nell’oligonucleotide., Non solo consente la determinazione del peso molecolare degli oligonucleotidi, ma anche in modo diretto o indiretto, la determinazione delle loro sequenze.
Software per l’analisi dei dati spettrometrici di massa
SimGlycan® predice la struttura dei glicani e dei glicopeptidi a partire dai dati MS / MS acquisiti dalla spettrometria di massa, facilitando gli studi di glicosilazione e modifica post-traslazionale. SimGlycan ® accetta i profili MS sperimentali, sia dei glicopeptidi che dei glicani rilasciati, li abbina con un proprio database e genera un elenco di strutture probabili., Il software supporta anche l’analisi dei dati di spettrometria di massa a più stadi che consente la delucidazione strutturale e l’identificazione dei percorsi di frammentazione.
SimLipid è un innovativo strumento di caratterizzazione lipidica che consente la delucidazione strutturale di lipidi sconosciuti utilizzando dati MS / MS. Il software analizza i dati spettrometrici di massa lipidica per caratterizzare e profilare i lipidi. SimLipid può anche annotare gli spettri di massa con le strutture lipidiche identificate usando le abbreviazioni.